Sequencing techniques - that’s a good start
Paulina Smaruj
Wiceprezes, Koło Naukowe Biologii Molekularnej UW
Sekwencjonowanie stało się codziennością współczesnej biologii. Techniki sekwencjonowania wykorzystują metody biochemiczne do określenie właściwej kolejności nukleotydów w makromolekułach DNA. Wynikiem sekwencjonowania jest ciąg znaków ze zbioru {A, C, T, G, N}, gdzie N oznacza nukleotyd, co do którego nie mamy pewności. Pierwszą metodą była technika Sangera stworzona w 1977 roku na Uniwersytecie w Cambridge, następną (i znaczenie rzadziej wspominaną) - Maxama z Uniwersytetu Harvarda (rok 1980).
Pan Sanger i dideoksynukleotydy
Sekwencjonowanie Sangera znane jest także pod nazwami: metoda terminacji łańcucha oraz metoda dideoksy. Idea w rzeczy samej bardzo prosta. Wyobraźmy sobie, że wybieramy jedną z nici DNA, która od teraz będzie pełnić dla nas funkcję matrycy. Następnie przeprowadzamy coś, co bardzo przypomina zwykłą reakcję PCR z jednym małym wyjątkiem - obok najzwyklejszych dNTP-ów dodajemy także jeden ddNTP (dideoksynukleotyd; jeden z: ddGTP, ddATP, ddTTP, ddCTP). Polimeraza DNA nie odróżnia pierwszych od drugich. Co jest nie tak z dideoksynukleotydami, że zasłużyły na inną nazwę? Otóż nie posiadają grupy hydroksylowej na końcu 3’, która jest kluczowa i niezbędna do przyłączenia kolejnego nukleotydu. Jaki jest skutek? Po inkorporacji ddNTP nie może zachodzić dalsza synteza - dochodzi do terminacji łańcucha.
Jak można się domyślić, po zakończeniu rekacji w probówce mamy mieszaninę molekuł DNA o znacznym zróżnicowaniu długości. Możemy teraz je ustawić od największego do najmniejszego wykorzystując elektroforezę DNA, a następnie zobrazować.
Pierwsze sekwencjonowania techniką Sangera wymagały przeprowadzenia czterech niezależnych rekacji na tej samej matrycy - w każdej dodano inny dideoksynukleotyd (Ryc.1). Następnie odczytywano sekwencję analizując żel elektroforetyczny od najkrótszych fragmentów do najdłuższych postępując według zasady: jeżeli prążek jest w ścieżce będącej wynikiem rozdziału mieszaniny, do której dodaliśmy np. ddGTP, to terminacja zakończyła się na G, więc kolejnym znakiem w sekwencji będzie „G”.
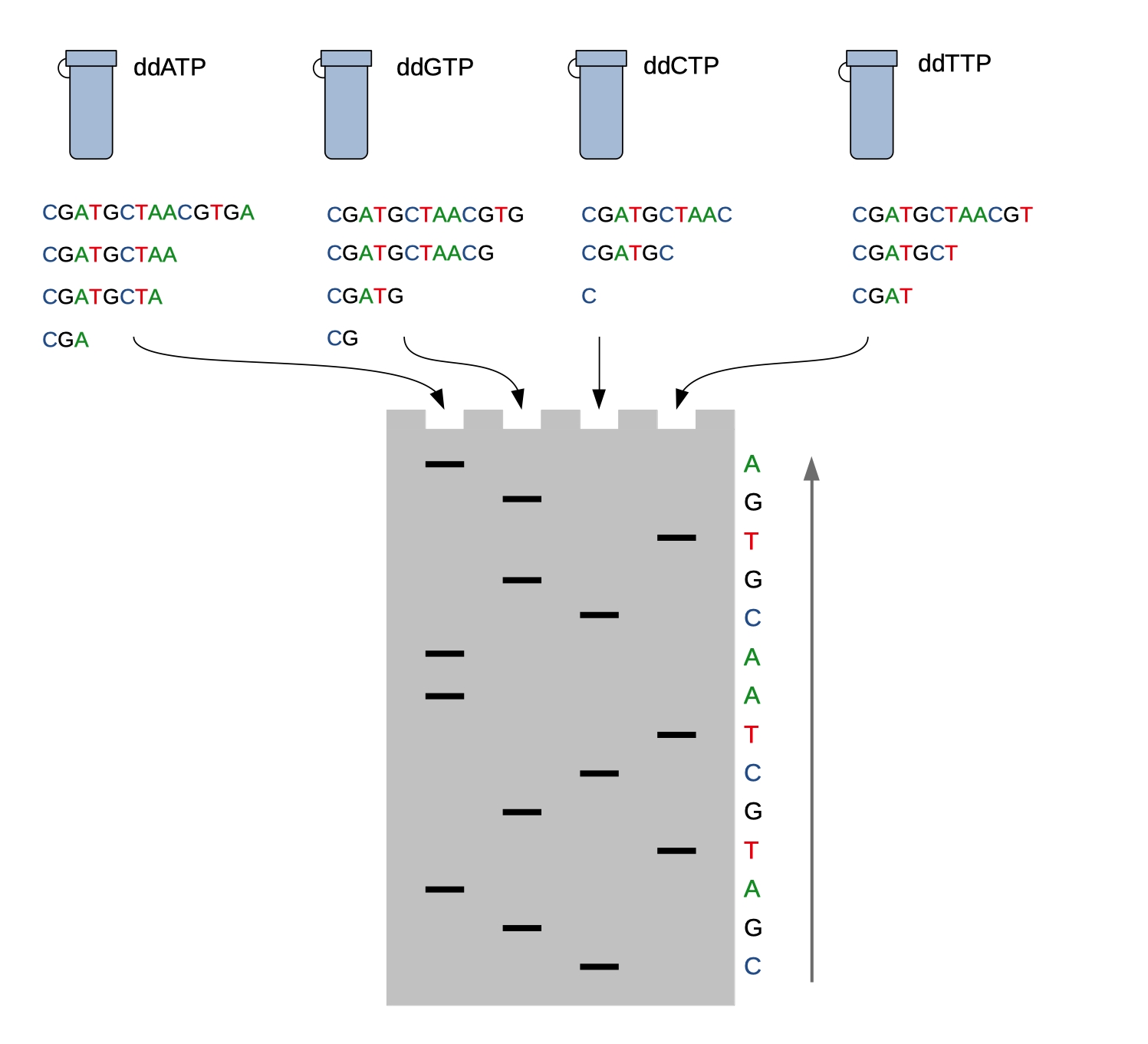
Ryc.1. Technika sekwencjonowania Sangera. Przeprowadzając 4 niezależne reakcje z dodatkiem jednego z dideoksynukleotydów można wydedukować sekwencję fragmentu DNA ze wzoru prążków po rozdziale elektoforetycznym.
W latach 90. znacznie zautomatyzowano technikę Sangera - od teraz wystarczam jedna reakcja, żeby określić sekwencję. Jak tego dokonano? Zmieniono metodę znakowania ddNTP - każdy dideoksynukleotyd otrzymał własny „kolor” (znakowanie fluorescencyjne). Mieszanina następnie rozdziela się techniką elektroforezy kapilarnej. Zautomatyzowanie procedury i detekcji pozwoliło na znaczne zmniejszenie czasu potrzebnego do określenia sekwencji - dzisiaj nikt nie wpatruje się żel elektroforetyczny, ale w ekran komputera.
Pan Maxam i niebezpieczne związki chemiczne
Technika Maxama-Gilberta inaczej określana jako metoda degradacji chemicznej jest współcześnie omawiana przede wszystkim ze względów historycznych. Idea też jest względnie prosta, chociaż podejście odmienne. Na początku musimy wyznakować nasz DNA - możemy zrobić to np. wykorzystując P32 (wyznakowane zostają końce 5’) (Ryc.2).
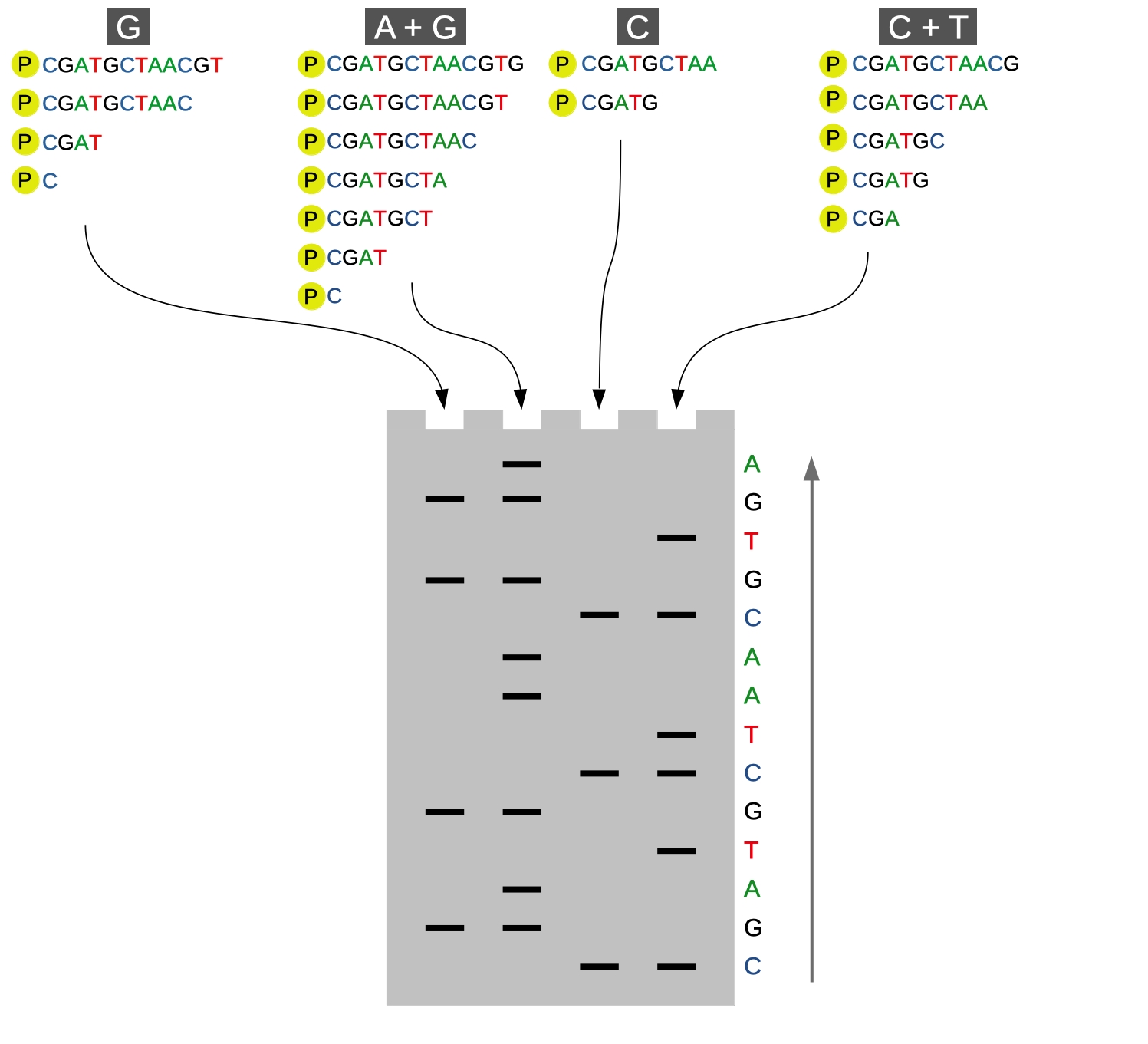
Ryc.2. Technika sekwencjonowania Maxama-Gilberta. Po dodaniu do DNA odpowiedniej mieszaniny związków chemicznych i przeprowadzeniu reakcji w określonych warunkach uzyskuje się mieszaninę fragmentów DNA, których rozdział elektoforetyczny umożliwia odczytanie sekwencji.
Tym razem będziemy traktować naszą próbkę DNA odczynnikami chemicznymi, które generują pęknięcia zależnie od tego, jaki nukleotyd jest obok. Przeprowadzamy równolegle 4 reakcje: C, T+C, G, A+G. W reakcji określanej jako „C” dojdzie do przecięcia obok cytozyny, z kolei w „T+C” - w miejscach, gdzie znajdzie się tymina lub cytozyna. Następnie przeprowadzamy elektroforezę, obrazujemy i odczytujemy sekwencję w sposób podobny jak w technice Sangera - tutaj jednak, jeżeli mamy prążek w próbce „A+G”, a nie mamy na tej samej wysokości prążka w „G”, wnioskujemy, że w kolejnym nukleotydem sekwencji jest adenina.
Pierwsza generacja technik sekwencjonowania była niesamowitym przełomem w biologii, choć na szybkie i wysokoprzepustowe techniki musieliśmy poczekać jeszcze kilka dekad - szczegóły w następnym artykule.
Bibliografia: