Igor Kłykociński
Skarbnik, Koło Naukowe Biologii Molekularnej UW
Na początku przypomnijmy sobie razem podstawowe terminy:
Locus (lm. loci) – miejsce na chromosomie zajmowane przez dany gen
Allel – alternatywna forma danego genu, allele danej pary zajmują to samo locus na chromosomach homologicznych
Genotyp – zespół genów danego organizmu
Fenotyp – zespół cech ujawniających się u danego organizmu, wynikający z genotypu oraz wpływu środowiska
Homozygota – organizm posiadający dwa identyczne allele w danym locus
Heterozygota – organizm posiadający dwa różne allele w danym locus
Allel dominujący – fenotyp przez niego kodowany ujawnia się u heterozygoty
Allel recesywny – fenotyp przez niego kodowany nie ujawni się u heterozygoty
Genetyka jako gałąź nauki powstała dzięki doświadczeniom Grzegorza Mendla. Wybrał on do badań groch zwyczajny (Pisum sativum). Następnie rozmnażał rośliny, by sprawdzić, czy dana cecha utrzymuje się w kolejnych pokoleniach. Po upewnieniu się, że wybrane w ten sposób rośliny należą do czystych linii, przystąpił do właściwego eksperymentu. Mendel wybrał rośliny o cechach (fenotypach), łatwych do odróżnienia, czyli np. barwa kwiatu (purpurowa lub biała), kolor nasion (żółty lub zielony), wysokość pędu (wysoki lub niski). W poniższym przykładzie omówimy wariant dotyczący wysokości pędu.
Pokolenie P (Parentes) – organizmy rodzicielskie należące do czystych linii
Pokolenie F1 (Fili 1) – organizmy powstałe ze skrzyżowania organizmów z pokolenia P
Pokolenie F2 (Fili 2) – organizmy powstałe ze skrzyżowania organizmów z pokolenia F1
T – allel dominujący
t – allel recesywny
P: TT x tt
F1: Tt
F2: TT Tt Tt tt
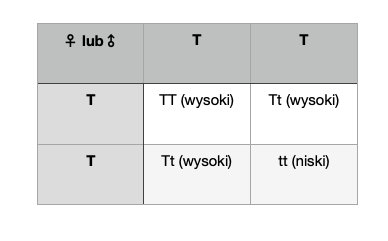
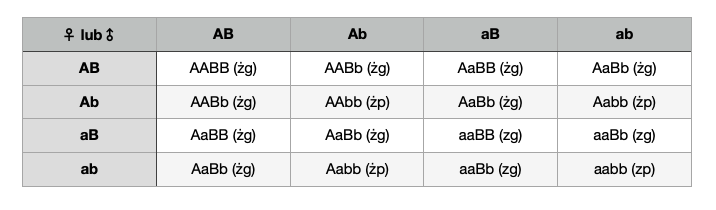
Z przeprowadzonego doświadczenia wynika, że organizmy (F1) powstałe ze skrzyżowania homozygoty dominującej i recesywnej (P), będą zawsze heterozygotami, a ich fenotyp będzie tożsamy z fenotypem homozygoty dominującej. Natomiast organizmy (F2), które otrzymano krzyżując ze sobą organizmy z pokolenia F1, będą miały genotypy: TT, Tt, tt (1:2:1), a fenotypy wysoki i niski (3:1). By w prosty sposób przewidzieć genotypy i fenotypy organizmów potomnych można wykonać krzyżówkę genetyczną w formie szachownicy Punnetta (w tym przykładzie wykonaliśmy ją dla heterozygot z pokolenia F1). Oprócz omówionej powyżej krzyżówki jednogenowej można wykonać również krzyżówkę dwugenową, polegającą na zbadaniu wzajemnego dziedziczenia genów z różnych loci. Przykładem takiego doświadczenia będzie skrzyżowanie grochu o fenotypach nasion żółtych lub zielonych (allele A i a) oraz nasion gładkich lub pomarszczonych (allele B i b). Otrzymane pokolenie F1 (AaBb), będzie w 100% składało się z heterozygot dominujących pod względem obydwu cech. Produkować zaś będzie gamety AB, Ab, aB, ab. Pokolenie następne, czyli F2, będzie złożone z organizmów o 9 różnych genotypach, dającymi 4 fenotypy nasion: żółte gładkie, żółte pomarszczone, zielone gładkie, zielone pomarszczone (9:3:3:1).
Na podstawie swoich doświadczeń Mendel sformułował dwa podstawowe prawa:
Prawo segregacji alleli (zwane również prawem czystości gamet), według którego w czasie mejozy allele z każdego locus rozdzielają się, wchodząc do różnych gamet. Innymi słowy, w jednej gamecie może się znaleźć tylko jeden allel z danej pary.
Prawo niezależnej segregacji cech (inaczej prawo niezależnego dziedziczenia), w myśl którego allele należące do danej pary genów segregują się (dziedziczą), w sposób niezależny od alleli należących do innej pary. To prawo nie jest spełnione dla genów sprzężonych, czyli takich, których loci znajdują się w bliskiej odległości od siebie na tej samej parze chromosomów homologicznych. Sprzężenie genów można wykazać przy pomocy krzyżówki testowej dwugenowej, w której skojarzymy heterozygotę (AaBb) i homozygotę recesywną (aabb). Wówczas, jeśli geny są sprzężone, to w pokoleniu potomnym fenotypy takie same, jak u rodziców, będą się pojawiały z większą częstotliwością niż inne (w genach niesprzężonych proporcje fenotypów powinny być równe).
Organizmy o fenotypach, które pojawią się jako nowe (nie występują u rodziców), to rekombinanty. Sumując liczbę otrzymanych rekombinantów, a następnie dzieląc ją przez całkowitą liczbę potomstwa, można określić częstość procesu crossing-over.
Bibliografia:
- Biologia, Eldra Pearl Solomon, Linda R. Berg, Diana W. Martin (str. 193 – 215)
- Pea (Pisum sativum L.) in Biology prior and after Mendel’s Discovery, Petr SMÝKAL